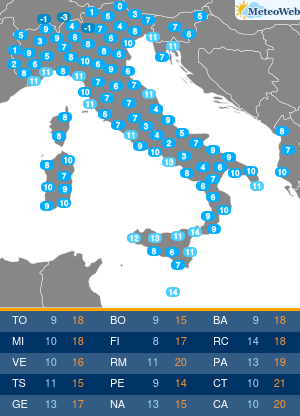
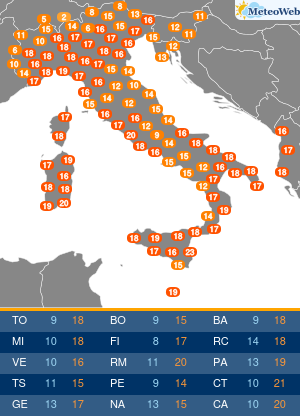
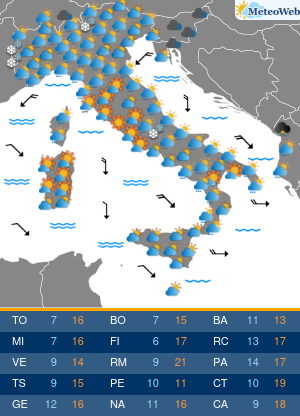
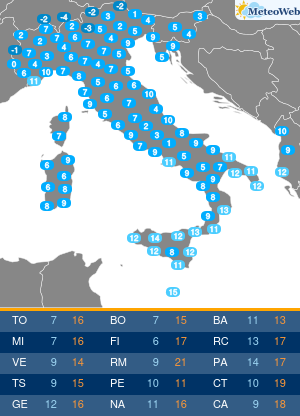
Dopo il primo successo ottenuto il mese scorso con il sequenziamento sul campo di una rana affine alla specie Amietia delalandi (un anfibio diffuso in alcune zone dell’Africa orientale) per il 96%, i ricercatori di Kabobo Expedition – spedizione di esplorazione biologica organizzata dal Muse di Trento in collaborazione con Universita’ di Verona e Wildlife Conservation Society – hanno sequenziato altri sei organismi viventi. Si tratta di due rettili, due anfibi e due mammiferi, un pangolino e una piccola antilope di foresta. Massimo Delledonne, professore dell’Universita’ di Verona e Anita Rodriguez Prieto, ricercatrice del Muse di Trento hanno estratto, purificato e amplificato il Dna degli animali nel laboratorio da campo attrezzato nella foresta del massiccio di Kabobo, lungo le sponde del Lago Tanganika.
“La tecnologia sviluppata da Oxford Nanopore Technologies che abbiamo utilizzato in questa spedizione – spiega Delledonne – ci ha permesso per la prima volta di caratterizzare le basi genetiche di specie selvatiche direttamente sul campo e completamente offline, in un momento in cui la scienza e’ sempre piu’ dipendente da internet”. I risultati ottenuti sono stati confrontati con le sequenze depositate nella banca dati GenBank. Il blast, ovvero il confronto delle nuove sequenze con quelle presenti nella banca dati, fornisce una prima importante indicazione per capire se si tratta di specie nuove alla scienza o per confermare l’identita’ di un organismo. Nel caso del pangolino arboricolo e della rana arboricola, il Dna ha mostrato una tale diversita’ con quello delle specie depositate nel database da far pensare che, se non nuove, queste siano comunque sostanzialmente diverse dalle altre, confermando quindi la singolarita’ della storia evolutiva di queste foreste. Anche nel caso dell’antilope, il blast indica che la specie potrebbe appartenere a un gruppo non ancora documentato nella banca dati o addirittura essere nuova.